En estudios de salud pública y epidemiología relacionados con enfermedades causadas por los virus y las bacterias, es importante contar con herramientas de laboratorio que permitan la identificación de la especie del agente patógeno responsable de la enfermedad. La detección e identificación de la especie bacteriana causante de una enfermedad, junto con otro tipo de evidencia colectada por el personal de salud que atiende a un paciente, como los síntomas que este presenta, permite obtener un diagnóstico correcto.
El diagnóstico acertado de una enfermedad infecciosa permite iniciar el tratamiento adecuado del paciente y la toma de decisiones para prevenir la dispersión de la enfermedad entre la población. En este artículo te presentamos un panorama general de cómo han progresado las técnicas de laboratorio de microbiología utilizadas para detectar e identificar las bacterias patógenas del ser humano.
El microscopio y el cultivo, las bases para la identificación bacteriana
Un grupo de herramientas de laboratorio empleadas en la identificación de una especie bacteriana se basan en el estudio de las características fenotípicas, con el que se analizan aspectos microscópicos morfológicos del patógeno, o bien propiedades bioquímicas como la capacidad para crecer en medios de cultivo que contienen nutrimentos particulares. La incorporación del microscopio para la observación directa de bacterias en muestras obtenidas del paciente proveniente de piel, sangre y cabello o de secreciones como saliva y orina, fue la primera herramienta del laboratorio de microbiología para la detección de bacterias patógenas.
La primera observación de una bacteria al microscopio la hizo el holandés Antony van Leeuwenhoek en 1673, que junto con la observación de hongos por el inglés Robert Hooke en 1665, marcan los inicios de la microbiología como ciencia moderna. El microscopio permitió el reconocimiento de distintas especies microbianas con base en la forma de sus células y sus estructuras de movilidad y adhesión asociadas, como fimbrias y flagelos, iniciando así la clasificación bacteriana para distinguir entre especies.
La observación de las bacterias directamente en las muestras clínicas de humanos, se optimizó empleando tratamientos químicos que generaban distintos tipos de coloración, permitiendo así contrastar mejor a la bacteria en el tejido e identificar grupos de bacterias específicas. Entre los ensayos de tinción de bacterias para su observación al microscopio, destaca la denominada tinción de Gram, desarrollada por el bacteriólogo danés Hans Christian Gram en 1884, para observar bacterias en tejido pulmonar de pacientes que habían muerto a causa de neumonía.
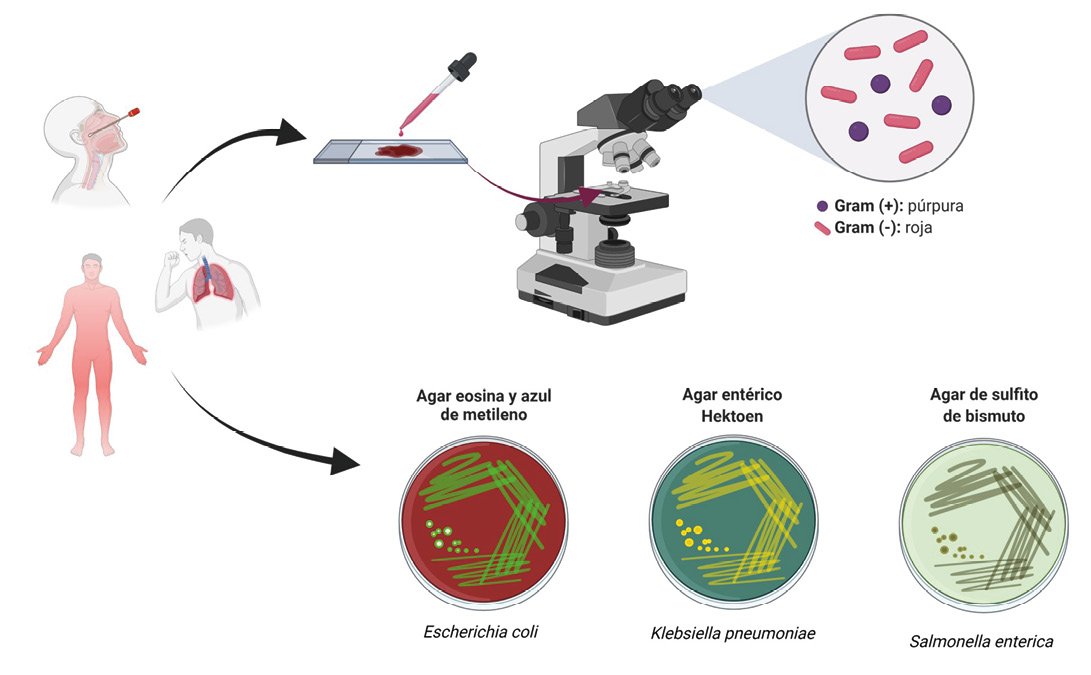
Entre las primeras herramientas empleadas para la identificación de bacterias patógenas y el diagnóstico de enfermedades causadas por estas, se encuentran el análisis microscópico de muestras clínicas tomadas al paciente y la tinción con colorantes específicos, como el caso de la tinción de Gram, así como el cultivo bacteriano en medios selectivos. Imagen elaborada con Biorender.com.
Dicha tinción revela diferencias en la estructura de la pared celular de las bacterias. Este ensayo dividió a las bacterias en dos grandes grupos, las que respondían al tratamiento químico y se podían observar teñidas al microscopio con una coloración azul-violeta, denominadas Gram-positivas, y las que no respondían al tratamiento presentando una coloración roja, denominadas Gram-negativas. A la fecha, esta clasificación se sigue empleando como criterio inicial de diagnóstico en enfermedades infecciosas. A partir del uso del microscopio, la identificación de bacterias patógenas avanzó de la mano de la determinación de especies para su clasificación biológica a nivel general.
Una herramienta de diagnóstico de laboratorio posterior al uso del microscopio fue el aislamiento de la bacteria procedente de los tejidos y fluidos del individuo enfermo, y su crecimiento en medios de cultivo adecuados. Aunque no existe un consenso, se atribuye al inglés Joseph Lister el primer cultivo puro de una bacteria entre 1877-1878, al realizar diluciones de leche para aislar a la bacteria responsable de la producción de ácido láctico y el cuajo de la leche. Microbiólogos como el francés Louis Pasteur ya habían elaborado medios de cultivo líquidos; pero es en el laboratorio del alemán Robert Koch en donde se prepara, entre 1881 y 1882, el medio de cultivo sólido para obtener cultivos puros de bacterias. El medio de cultivo se desarrolla gracias a Fanny Eilshemius, quien le sugirió a su esposo Walther Hesse, colaborador de Koch, la inclusión del agar en la formulación. En la actualidad existe una gran variedad de medios de cultivo específicos, que permite distinguir fácilmente a distintas bacterias patógenas mediante cambios de color y formas de crecimiento, entre otras características.
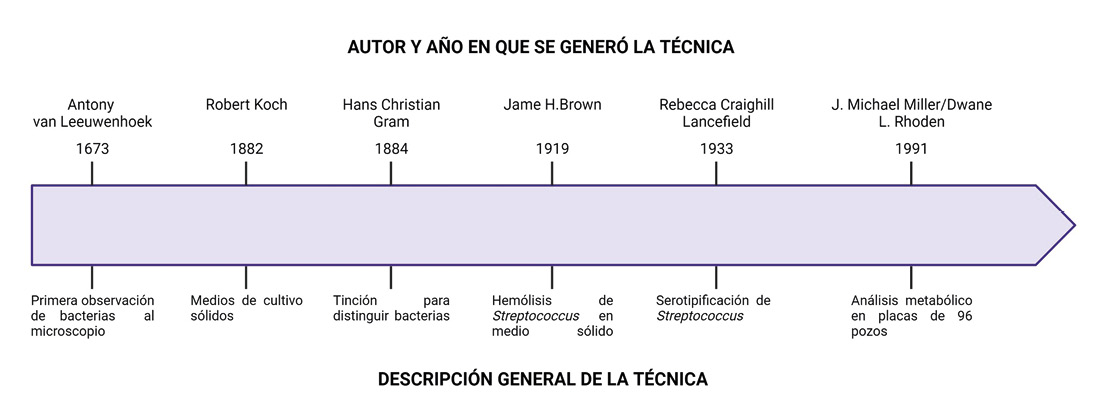 Línea de tiempo del surgimiento de algunas de las herramientas de diagnóstico fenotípico relevantes. El tiempo no está en escala. Imagen elaborada con Biorender.com.
Línea de tiempo del surgimiento de algunas de las herramientas de diagnóstico fenotípico relevantes. El tiempo no está en escala. Imagen elaborada con Biorender.com.
Pruebas bioquímicas y metabólicas en la identificación de bacterias infecciosas
Una vez que se pudo realizar el cultivo puro de bacterias patógenas, se conocieron sus diferencias en la capacidad para producir sustancias químicas y para emplear enzimas que les permitían utilizar o procesar distintos tipos de compuestos químicos adicionados al medio. Así se marcó un tercer paso relevante en el diagnóstico microbiológico, la identificación de moléculas y enzimas, característicos de cada especie bacteriana. Por ejemplo, se encontró que algunas bacterias eran capaces de destruir glóbulos rojos en un medio de cultivo al que se le había añadido sangre, creando un cambio de color en el medio. Al proceso de destrucción de los glóbulos rojos se le denominó hemólisis y la observación permitió diseñar un medio de cultivo para el diagnóstico de este tipo de bacterias.
Aunque no se tiene la certeza sobre quién diseñó el medio de cultivo con sangre, los estudios de James H. Brown sobre la capacidad hemolítica de bacterias del género Streptococcus realizados entre 1915 y 1919, figuran entre los primeros que usan esta herramienta de cultivo para clasificar bacterias patógenas. La capacidad de metabolizar distintos azúcares para la producción de alcohol, denominada fermentación, fue también incorporada como prueba diagnóstica dentro de este grupo de herramientas.
Una derivación técnica relativamente reciente, originada de la capacidad de cultivo bacteriano y de las diferencias en las capacidades bioquímicas y metabólicas de las distintas especies, fue la sustitución del medio de cultivo sólido en placas de Petri por placas de plástico con 96 pozos, que contienen cada uno distintas moléculas orgánicas como carbohidratos, aminoácidos y ácidos orgánicos, entre otros. La bacteria de interés aislada del paciente se inocula en cada uno de los pozos en medio líquido y se determina su capacidad para metabolizar y crecer en presencia de cada una de las moléculas de prueba en los pozos de la placa. Esta técnica, propuesta en 1991 por los estadounidenses J. Michael Miller y Dwane L. Rhoden para la identificación de bacterias patógenas, permitió la evaluación de múltiples condiciones de cultivo en un solo ensayo, así como la posterior lectura automática mediante un equipo de cómputo de los resultados.
En la segunda mitad del siglo pasado se incorporó la espectrometría de masas para la identificación de biomoléculas presentes en las células bacterias. En 1973, Henk L. C. Meuzelaar y Piet G. Kistemaker, observaron por primera vez patrones específicos de señales de moléculas orgánicas ionizadas producidas por el calentamiento de bacterias de los géneros Neisseria y Leptospira. Dos años más tarde, John P. Anhalt y Catherine Clarke Fenselau, propusieron la espectrometría de masas como herramienta para la identificación bacteriana. La espectrometría de masas experimentó un rápido desarrollo en décadas posteriores y, actualmente, es capaz de analizar moléculas como proteínas, ADN y lípidos, que son ionizadas mediante técnicas especiales aun en las células intactas, detectando patrones específicos inclusive entre cepas distintas de la misma especie bacteriana, lo que la hizo una herramienta eficaz en el diagnóstico microbiológico.
El sistema inmune ayudó en la identificación de bacterias patógenas
Con los estudios del sistema inmune en seres humanos y distintos animales de laboratorio, como ratones y conejos, se conoció un tipo de proteínas muy particular denominadas anticuerpos, que se producen para identificar agentes extraños dentro del cuerpo, llamados genéricamente antígenos. Los anticuerpos reconocen y se unen a moléculas de la superficie de las bacterias, como azúcares, lípidos y proteínas, que permiten su identificación como agentes extraños al cuerpo y su posterior destrucción por el sistema inmune. Este conocimiento permitió el nacimiento de una nueva herramienta en el diagnóstico de enfermedades bacterianas, la serotipificación.
El trabajo pionero de la estadounidense Rebecca Craighill Lancefield, quien estudió las moléculas antigénicas del género Streptococcus entre 1920 y 1933, le permitió su división en distintos serotipos o serogrupos, diferenciando los prevalentes y más virulentos en humanos, de los que se aislaban de animales. Se crea así un grupo de sueros con distintos anticuerpos que sirven para identificar diferentes componentes químicos de la superficie de un patógeno particular. En esta técnica se utilizan anticuerpos dirigidos contra distintas especies o variantes de una misma especie bacteriana, estrategia denominada de manera general como subtipificación.
La subtipificación bacteriana fue un evento relevante en epidemiología y salud pública en general, ya que permitió distinguir aquellas variantes que causan una enfermedad más grave de aquellas que presentan solo síntomas leves de la enfermedad, o que entran al cuerpo sin ser detectadas por la falta de síntomas detectables por los médicos. A la fecha, existen diversas variantes de identificación de bacterias patógenas basadas en la aplicación de anticuerpos, denominadas genéricamente inmunoensayos. Un ensayo común es el ELISA, derivado de sus siglas en inglés, Enzyme-Linked Immuno Sorbent Assay (ensayo por inmunoadsorción ligado a enzimas).
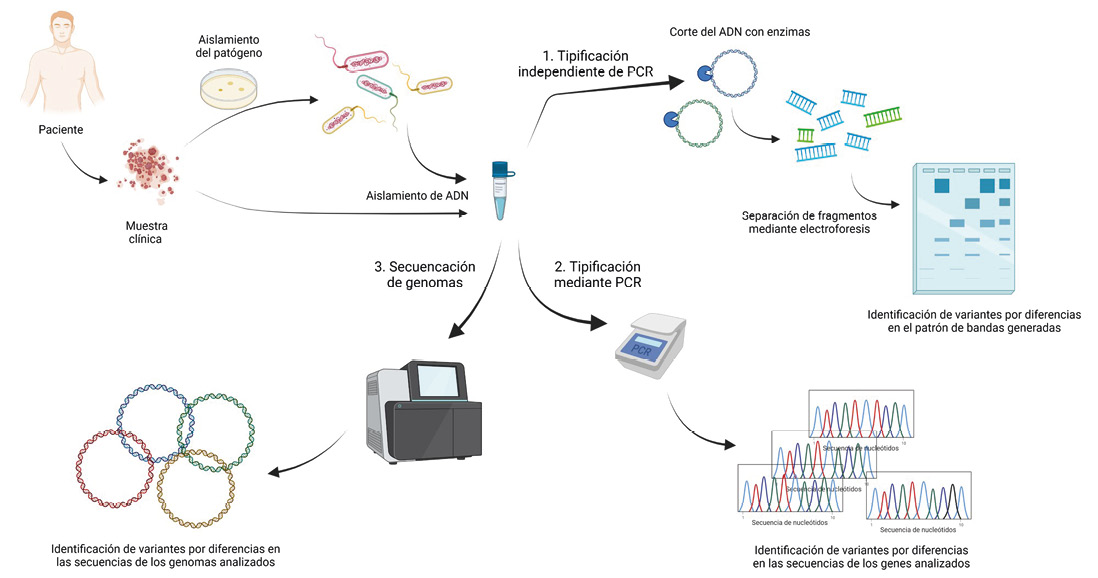 Ejemplos de tres técnicas de tipificación genético molecular de bacterias patógenas. Algunas se basan en el ensayo de Reacción en cadena de la Polimerasa (PCR, por sus siglas en inglés) y otras no. La herramienta más actual en este grupo es la secuenciación de genomas completos. Imagen elaborada con Biorender.com.
Ejemplos de tres técnicas de tipificación genético molecular de bacterias patógenas. Algunas se basan en el ensayo de Reacción en cadena de la Polimerasa (PCR, por sus siglas en inglés) y otras no. La herramienta más actual en este grupo es la secuenciación de genomas completos. Imagen elaborada con Biorender.com.
El diagnóstico y la identificación genético-molecular
A partir de las últimas dos décadas del siglo pasado, se inició la detección e identificación bacteriana mediante el análisis de su material genético o ácido desoxirribonucleico (ADN), naciendo así las denominadas técnicas de identificación y tipificación genético-molecular. Desde entonces, a la fecha, se han diseñado y abandonado un gran número de este tipo de técnicas por diferentes grupos de investigación en el mundo, que por razones de espacio sería imposible mencionarlas en este artículo, pero te describimos las principales.
Durante este periodo se han abordado distintas clasificaciones de las técnicas genético-moleculares de tipificación. Por ejemplo, podían dividirse en dos grupos, de acuerdo con las características generales de la metodología empleada, diferenciándose en aquellas basadas en «patrones de bandeo» del ADN y las basadas en secuencias de ADN. El término «patrones de bandeo» se refiere a que las diferencias entre las bacterias analizadas se establecen con base en fragmentos de ADN de distinto tamaño molecular que se pueden visualizar mediante métodos de rutina en un laboratorio de biología molecular. La técnica de patrones de bandeo que ha sido considerada un estándar de oro para la y subtipificación bacteriana, es la Electroforesis en Geles de Campo Pulsado (Pulse Field Gel Electrophoresis, PFGE), introducida por David C. Schwartz y Charles R. Cantor en 1984.
Otra clasificación de las técnicas de identificación genético molecular, distingue entre aquellas basadas en la Reacción en Cadena de la Polimerasa (Polymerase Chain Reaction, PCR) y las no basadas en esta herramienta. La técnica de PCR fue descrita por Kary Mullis y sus colegas en 1986. Quizá en los últimos meses has oído hablar bastante de esta técnica por su relevancia para el diagnóstico de COVID-19 para detectar al virus SARS-CoV-2, y debido a la gran cantidad de información que se ha generado al respecto (no abordaremos en detalle su fundamento y aplicación).
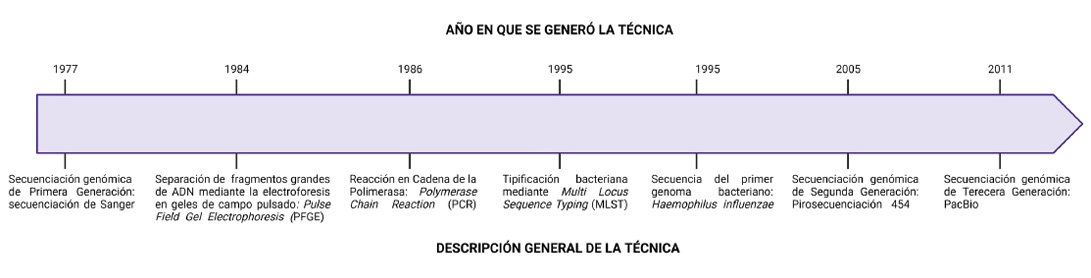
Línea de tiempo del surgimiento de algunas de las herramientas de diagnóstico genético molecular relevantes. El tiempo no está en escala. Imagen elaborada con Biorender.com.
En el análisis de secuencias de ADN para la tipificación bacteriana, se utilizaron inicialmente uno o dos genes para identificar a la especie de interés. Posteriormente, un equipo de científicos coordinados por Martin C. J. Maiden incorporó en 1988 el análisis de la secuencia de siete genes, en una técnica a la que se designó por sus siglas en inglés como MLST (Multi Locus Sequence Typing). Por sus características, esta técnica permitió el análisis de poblaciones de bacterias de la misma especie, distinguiendo de una manera muy precisa aquellas bacterias relacionadas con brotes de infecciones en una población o grupo de individuos, con las bacterias de la misma especie que se encontraban presentes en el área del brote, pero que no tenían relación con este.
Lo que en la actualidad se considera la herramienta más poderosa de identificación y tipificación para el diagnóstico surgida en los últimos años, ha sido sin duda alguna la obtención y el análisis de la Secuencia de Nucleótidos del Genoma completo de las bacterias (por sus siglas en inglés Whole Genome Sequencing, WGS). Esto ha sido posible debido al desarrollo de técnicas de secuenciación masiva que permiten la obtención de millones de datos sobre la secuencia de un genoma, tan solo en unas horas o pocos días, dependiendo de la técnica de secuenciación empleada y del tamaño del genoma analizado.
Debido a que las técnicas de secuenciación masiva generan fragmentos pequeños de ADN, se requiere de herramientas de cómputo para «ensamblar» el genoma en el orden correcto. Para que puedas darte una idea de este proceso, piensa en un rompecabezas de un gran número de piezas que quieras ensamblar para conocer la imágen completa. Las herramientas de cómputo para el ensamble y análisis de secuencias de genomas bacterianos completos, son toda un área de especialización denominada Bioinformática, que también se aplica para el análisis de otras moléculas biológicas complejas como proteínas, carbohidratos, lípidos y ácido ribonucleico (ARN), con algunas diferencias específicas para cada caso. Estas herramientas incluyen el diseño de programas informáticos o software especializado.
Herramientas de diagnóstico cada vez más simples
Aunque en la actualidad el diagnóstico y análisis epidemiológico mediante la comparación de genomas completos es una labor compleja y altamente especializada, cada año las herramientas para desarrollar dicha tarea se han ido simplificando y realizando mediante sitios de internet que facilitan el análisis y la interpretación de los datos. Es de esperar que en un futuro muy cercano tanto la obtención de genomas bacterianos como su análisis, sean accesibles en costo para todo el interesado y sin la necesidad de tener un gran conocimiento para el manejo de programas de computadora especializados.
La secuenciación de genomas completos para el diagnóstico y la tipificación bacteriana ya es una técnica rutinaria y establecida como requisito en varios países del primer mundo y que en el corto tiempo se extenderá a la mayoría de los países. Sin duda alguna la pandemia por COVID-19 por la que todavía estamos atravesando, ha mostrado la relevancia de la secuenciación de genomas de cualquier patógeno como la herramienta que llegó para quedarse por un largo tiempo en el laboratorio de diagnóstico microbiológico.
Otro avance significativo en las últimas décadas es el desarrollo de kits de diagnóstico portátiles y de fácil manejo, de manera similar a las pruebas de glucosa y de embarazo, que hacen uso de algunos de los fundamentos mencionados anteriormente. Es cada vez más común la presencia de este tipo de pruebas de diagnóstico que se pueden adquirir en las farmacias y realizar el ensayo en casa. Este desarrollo también se ha acelerado en los dos últimos años como consecuencia de la pandemia de COVID-19. Por motivos de espacio, ya no se detallan dichas aplicaciones, pero el lector encontrará mucha información al respecto navegando en la red.
Para Saber Más:
Balsalobre-Arenas, L. y Alarcón-Cavero, T. (2017). Diagnóstico rápido de las infecciones del tracto gastrointestinal por parásitos, virus y bacterias. Enfermedades Infecciosas y Microbiología Clínica, 35(6), 367-376. https://www.sciencedirect.com/science/article/pii/S0213005X17300228
Hervé, B. (2015). Nuevas tecnologías en diagnóstico microbiológico: automatización y algunas aplicaciones en identificación microbiana y estudio de susceptibilidad. Revista Médica Clínica Las Condes, 26(6), 753-763. https://www.sciencedirect.com/science/article/pii/S0716864015001510
Serrano, M.R.G., Escartín, N.L., Arriaza, M.M. y Díaz, J.C.R. (2019). Diagnóstico microbiológico de la bacteriemia y la fungemia: hemocultivos y métodos moleculares. Enfermedades Infecciosas y Microbiología Clínica, 37(5), 335-340. https://www.sciencedirect.com/science/article/abs/pii/S0213005X18300806
Vila, J., Gómez, M.D., Salavert, M. y Bosch J. (2017). Métodos de diagnóstico rápido en microbiología clínica: necesidades clínicas. Enfermedades Infecciosas y Microbiología Clínica, 35(1), 41-46. https://www.sciencedirect.com/science/article/abs/pii/S0213005X16303500
Elda Araceli Hernández Díaz. candidata a Doctora, Doctorado en Ciencias Biológicas en la Opción de Biotecnología Molecular, del Centro Multidisciplinario de Estudios en Biotecnología, Facultad de Medicina Veterinaria y Zootecnia de la UMSNH. Realizó sus estudios de Licenciatura en la Facultad de Quimicofarmacobiología-UMSNH y los estudios de Maestría en Ciencias de la Salud de la División de Estudios de Posgrado de la Facultad de Ciencias Médicas y Biológicas “Dr. Ignacio Chávez”-UMSNH. Su interés es en el análisis de virulencia y resistencia a antibióticos, tanto experimental como bioinformático, de bacterias patógenas de humano.
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
Gerardo Vázquez-Marrufo. Realizó la licenciatura en Químico Farmacéutico Biólogo (QFB) en Facultad de Química de la Universidad de Guanajuato y la Maestría en Ciencias en la misma institución. Obtuvó el Doctorado en Ciencias en la Unidad de Biotecnología e Ingeniería Genética de Plantas del CINVESTAV- IPN, Unidad Irapuato. Es Nivel III del Sistema Nacional de Investigadores (SNI). Ha publicado 46 artículos en revistas internacionales con índice de impacto. Le interesa la epidemiología molecular de bacterias patógenas de humano y el estudio de hongos silvestres con potencial biotecnológico.
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
