Relevancia del diagnóstico de COVID-19
Desde el inicio de la pandemia ocasionada por COVID-19, una de las prioridades ha sido elaborar pruebas o ensayos para el diagnóstico correcto y oportuno de dicha enfermedad causada por el virus SARS-CoV-2. El diagnóstico oportuno de COVID-19 es importante debido a que personas infectadas que todavía no desarrollan síntomas de la enfermedad y que no siguen las medidas de higiene, prevención y distanciamiento social adecuadas, son agentes de contagio y dispersión de la enfermedad. Además, en sus etapas iniciales, los síntomas de las infecciones provocadas por otros virus que atacan el sistema respiratorio son similares a los de COVID-19, por lo que, si el diagnóstico no es el correcto, un paciente puede ser internado en una unidad médica sin necesidad, o bien no recibir la atención y el tratamiento adecuados y resultar en un factor de contagio.
Aunque en distintas pruebas de diagnóstico clásicas o convencionales, es necesario aislar y cultivar al patógeno microbiano o partícula viral, a partir de una muestra clínica de saliva, orina, sangre, o cualquier tejido afectado por la infección, en las pruebas o ensayos de diagnóstico más recientes esto no es necesario, ya que pueden detectar al agente patógeno directamente en la muestra clínica.
Entre otras características, una prueba diagnóstica debe ser específica, sensible y reproducible. La especificidad implica que la prueba identifica únicamente al agente patógeno de interés, y no a otros patógenos similares. La sensibilidad indica la capacidad de la prueba para detectar al patógeno en las etapas iniciales de la infección, cuando su población dentro del paciente aún no ha alcanzado un gran número, algo que varía de patógeno a patógeno. Por último, la reproducibilidad se refiere al hecho de que los resultados positivos o negativos sean consistentes, independientemente del número de veces que se haga el ensayo y de quién y en dónde se realice.
Tipos de pruebas para el diagnóstico de COVID-19
El diagnóstico de una enfermedad infecto-contagiosa incluye diversas pruebas de laboratorio para la identificación de “huellas” bioquímicas o genéticas que puedan ser asociadas con un agente patógeno en particular. Las “huellas” bioquímicas detectan actividades enzimáticas o proteínas particulares, que distinguen a las capacidades metabólicas o características estructurales de un microorganismo particular. La detección de anticuerpos producidos por el sistema inmune del paciente en respuesta a la infección de un patógeno en particular, podría considerarse una variante de las “huellas” bioquímicas, aunque comúnmente se clasifica en la categoría de inmunodiagnóstico. Esta variante del diagnóstico se aplica a los virus y constituye la base de las denominadas pruebas rápidas.
En el caso de las “huellas” genéticas se busca detectar regiones específicas del material genético del patógeno. En bacterias y hongos, así como en otros patógenos, el blanco a localizar es una secuencia del ADN del microorganismo de interés. En relación a los virus, esta “huella” genética puede ser también un fragmento o región de ARN, debido a que hay virus, como el SARS-CoV-2, cuyo material genético es ARN y no ADN.
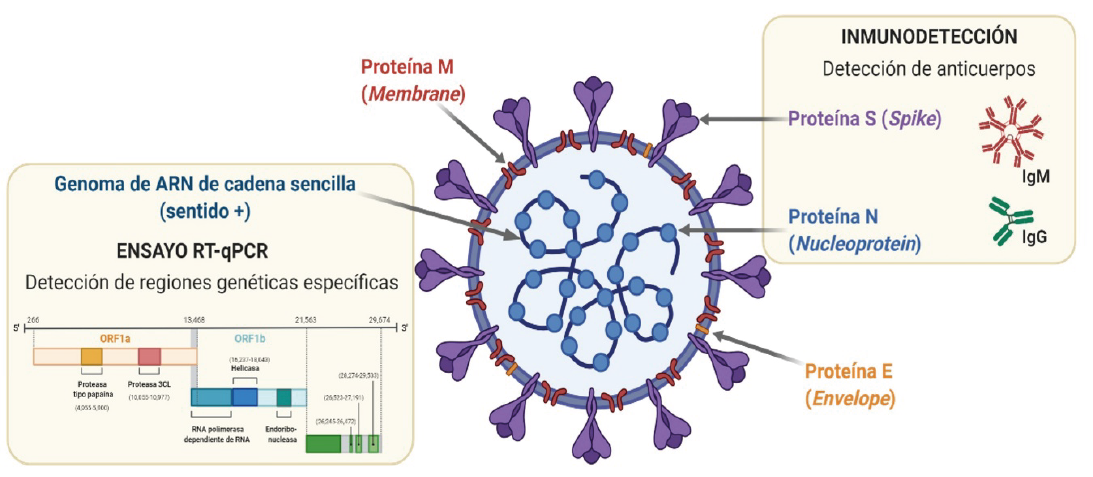
Estructura del virión del SARS-CoV-2. Se muestran las proteínas y el genoma de ARN, así como las pruebas de diagnóstico en que cada una de estas moléculas es empleada. Se indican en inglés los nombres de las proteínas de membrana (M), de espiga (S), de envoltura (E) y la nucleoproteína (N). Creada con BioRender.com.
Así, las pruebas de diagnóstico de COVID-19 pueden clasificarse en pruebas de inmunodiagnóstico, que detectan anticuerpos producidos por el sistema inmunológico del paciente para atacar al virus; y pruebas genéticas, que detectan regiones del ARN del SARS-CoV-2. Para realizar inmunodiagnóstico se parte de una muestra de sangre del paciente, en el caso de las pruebas genéticas, las muestras se obtienen principalmente introduciendo un hisopo en la nariz o boca, o bien a partir de saliva o esputo.
Pruebas de inmunodiagnóstico
En la mayoría de las pruebas y kits disponibles actualmente, se detectan los anticuerpos de las inmunoglobulinas IgM e IgG, producidos por el sistema inmune de la persona infectada. La proteína más empleada en estas pruebas es la S (spike=espiga), que forma parte de la cápside del virus. Puede utilizarse tanto la proteína completa, como algunos fragmentos de ésta, particularmente la secuencia de aminoácidos relacionada con el reconocimiento del receptor celular por el que el virus inicia el proceso de internalización en las células del paciente. Otra proteína utilizada es la N o nucleoproteína, que en el virión está asociada con el ARN.
En el inmunodiagnóstico comúnmente una señal de color, luminosa o fluorescente, indica que la proteína del virus usada en el ensayo detectó y se unió a los anticuerpos específicos contra el SARS-CoV-2 en el suero del paciente. El tiempo que consume el desarrollo de estas pruebas por los kits diseñados a la fecha, oscila entre los 15-120 min, característica a la cual deben su nombre genérico de pruebas rápidas, lo que constituye una de sus principales ventajas. No obstante, algunas pruebas de diagnóstico genético tienen tiempos de desarrollo similares.
Debido a que en estas pruebas, el diagnóstico positivo implica la detección de anticuerpos, su efectividad depende de que el paciente haya desarrollado una respuesta inmune efectiva contra el virus. Esto genera algunas desventajas para las pruebas mediante inmunodiagnóstico. Por una parte, el tiempo de incubación de la enfermedad es de 5 a 6 días, antes del desarrollo de una respuesta inmune, ocasionando que un portador del virus pre-asintomático genere un resultado negativo. Por otra parte, la intensidad de la respuesta inmune varía de una paciente a otro, y en algunos casos el desarrollo de anticuerpos contra el virus SARS-CoV-2 es tan bajo, que no pueden detectarse por las pruebas inmunológicas actuales.
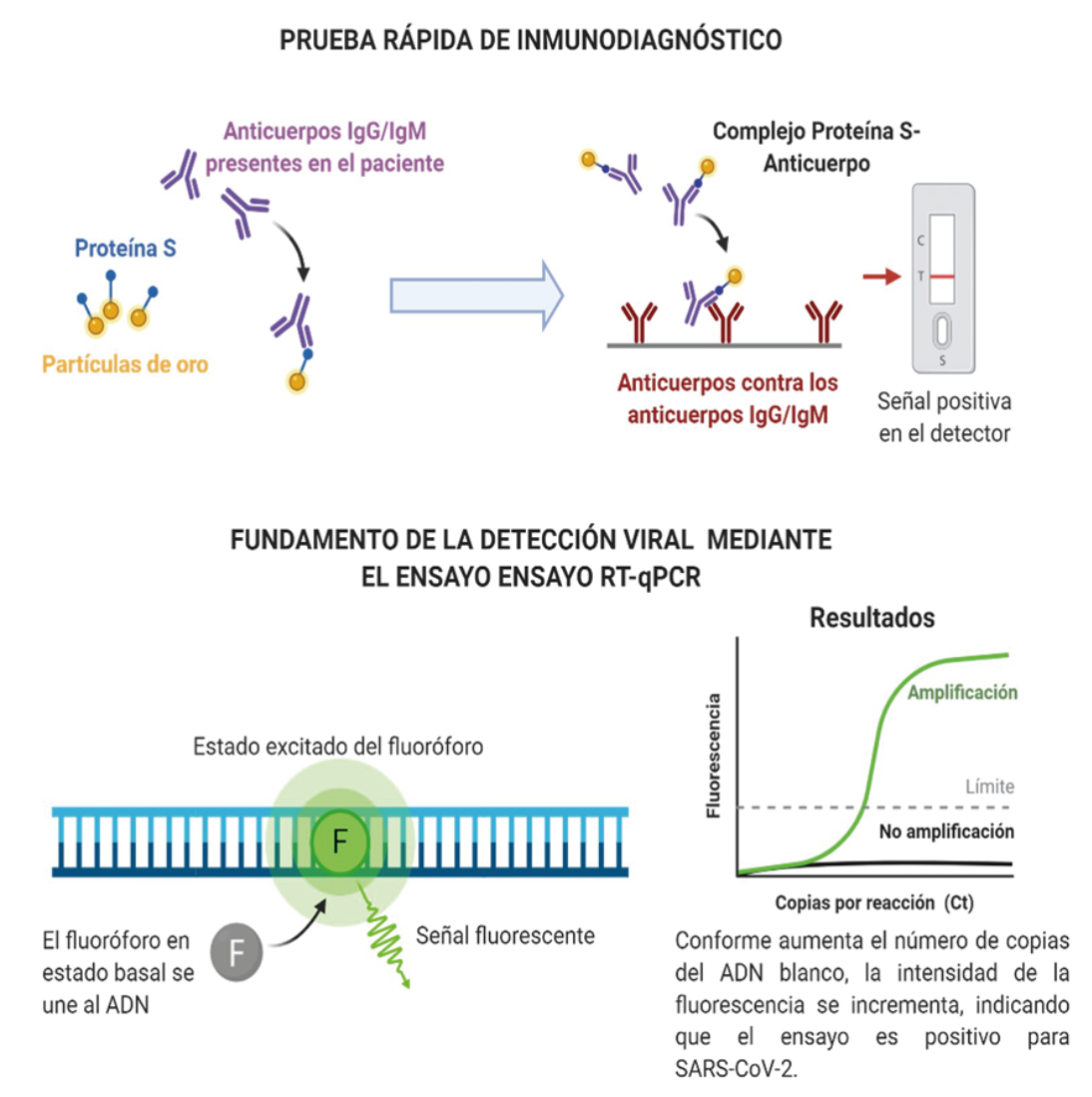 Fundamentos del inmunodiagnóstico y el ensayo de RT-qPCR para el diagnóstico de COVID-19. Creada con BioRender.com.
Fundamentos del inmunodiagnóstico y el ensayo de RT-qPCR para el diagnóstico de COVID-19. Creada con BioRender.com.
Pruebas de diagnóstico genético
La detección de regiones del ARN del virus SARS-CoV-2 es un ensayo de dos pasos que ofrece los mejores resultados de sensibilidad, especificidad y reproducibilidad. El primer paso es un proceso de transcripción reversa o inversa (RT, por sus siglas en inglés: Reverse Transcription), en el que a partir del ARN viral se genera el ADN complementario (cDNA, por sus siglas en inglés), mediante una enzima denominada transcriptasa reversa. El segundo paso es una amplificación en la que puede cuantificarse, en el momento en el que se van generando, el número de copias de la región genética de interés. Este paso se denomina Reacción en Cadena de la Polimerasa cuantitativa en tiempo real, abreviado como qPCR por sus siglas en inglés (quantitative Polymerase Chain Reaction). Así, la acronimia completa del ensayo en inglés es RT-qPCR. Este segundo paso en realidad es un proceso de tres etapas que se dan a diferentes temperaturas, generando millones de copias de la región de interés.
El tamaño del genoma del virus SARS-CoV-2 es de aproximadamente 30,000 bases, y el conocimiento de su secuencia completa permitió el diseño de ensayos para la detección de regiones ubicadas dentro de las estructuras genómicas denominadas Marcos de Lectura Abierta (Open Reading Frames, ORFs), ORF1a y ORF1b. También hay ensayos de RT-qPCR que detectan regiones dentro de los genes que codifican para las proteínas de envoltura E (envelope) y la nucleoproteína N. La detección de las copias generadas de la región de interés, también denominados productos de amplificación, se hace mediante una molécula fluorescente que incrementa la intensidad de la señal emitida conforme aumenta el número de copias generado. La especificidad para detectar una región genética particular depende de un par de secuencias cortas de ADN que reconocen los extremos de la región de interés, denominados iniciadores o primers, que delimitan la zona de copiado o amplificación. Estos componentes del ensayo también determinan en gran medida la detección específica del SARS-CoV-2, discriminándolo de otros virus que afectan el sistema respiratorio. Evitando así la generación de resultados falsos positivos. Con algunas variantes en relación a la región genómica de interés, este ensayo es el que utilizan todas las autoridades de salud en el mundo, por lo que su esquema general se muestra en la figura A.
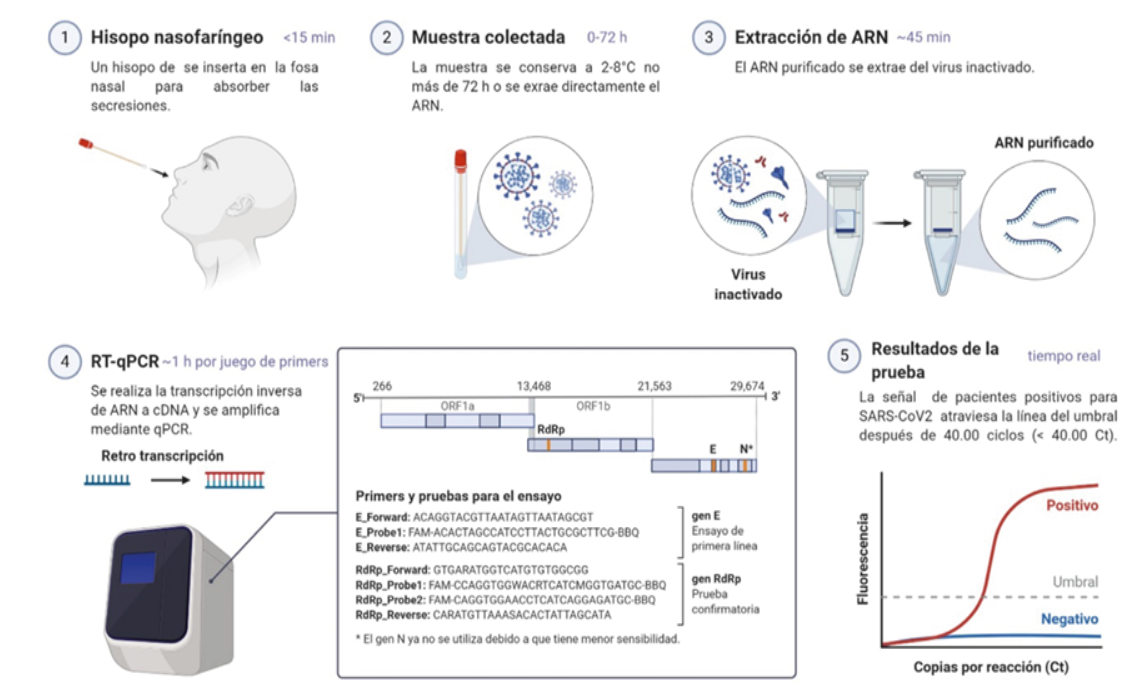
Figura A: Diagrama general de la prueba de RT-qPCR para el diagnóstico de COVID-19. Se muestra el orden de cada paso desde la obtención de la muestra clínica hasta la generación del resultado. Dentro del cuadro se muestran los iniciadores o primers y las regiones del genoma a la que va dirigido uno de varios protocolos diseñados. Creada con BioRender.com.
En el transcurso del desarrollo de la pandemia de COVID-19, se han diseñado una gran variedad de pruebas para la detección del ARN viral, empleando diversas metodologías de amplificación y detección que es imposible abarcar aquí. Entre estas pruebas puede mencionarse el ensayo de amplificación isotérmica mediada por bucle u horquilla (Loop-Mediated Isothermal Amplification, LAMP). A diferencia del ensayo RT-qPCR, el ensayo LAMP se desarrolla a una sola temperatura, y la interpretación de los resultados puede realizarse a simple vista. Estas dos ventajas del ensayo LAMP lo hacen más barato y fácil de desarrollar que el ensayo RT-qPCR, ya que no emplea el equipo costoso usado en este último. Actualmente ya existen kits comerciales de ensayo LAMP para la detección del SARS-CoV-2, y en algunos casos ya se está aplicando como una prueba rápida, cuyos resultados están disponibles en 45 minutos.
Esperamos haber dado una idea general que permita un mejor entendimiento sobre las bases de las pruebas de diagnóstico empleadas para detectar al virus SARS-CoV-2 en muestras clínicas. Contribuyendo así a tener una población mejor informada sobre un problema de salud pública mundial, que ha afectado todos los aspectos de nuestra vida diaria.
Saber Más:
Pinilla B.G., Cubillos K. y Rodríguez M. 2008. Bodas de plata de la reacción en cadena de la polimerasa (PCR). Nova, 6(9): 65-75.
https://revistas.unicolmayor.edu.co/index.php/nova/article/view/107
Marimón J.M. y Navarro-Marí J.M. 2017. Métodos de diagnóstico rápido de las infecciones respiratorias. Enfermedades Infecciosas y Microbiología Clínica, 35(2):108-115. https://www.sciencedirect.com/science/article/pii/S0213005X16303871
Gerardo Vázquez Marrufo, Profesor - Investigador del Centro Multidisciplinario de Estudios en Biotecnología, Facultad de Medicina Veterinaria y Zootecnia, Universidad Michoacana de San Nicolás de Hidalgo.
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
Ma. Soledad Vázquez Garcidueñas, Profesora - Investigadora de la División de Estudios de Posgrado, Facultad de Ciencias Médicas y Biológicas “Dr. Ignacio Chávez”, Universidad Michoacana de San Nicolás de Hidalgo.
Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
