
Un gen es una secuencia del ácido desoxirribonucleico (ADN) que ocupa un lugar determinado (locus) en un cromosoma con la información necesaria para definir el aspecto, estructura o función de un organismo; esta secuencia es formada por la unión de moléculas denominadas nucleótidos. La clonación de genes se refiere al copiado de una molécula única de ADN en una célula, para producir un gran número de células que contienen moléculas de ADN idénticas. Para ello se utilizan diversas herramientas que incluyen las “tijeras” que cortan el ADN y los vectores de clonación.
“Tijeras” que cortan el ADN
La primera herramienta que mencionaré pueden ser consideradas las “tijeras” que cortan el ADN; éstas son un tipo de enzimas que reconocen, se unen y cortan secuencias específicas de nucleótidos de 4, 6 o más pares de bases (pb) en el ADN de doble cadena; el descubrimiento de estas enzimas fue en las bacterias, realizado por Smith y Nathans en la década de 1970, científicos galardonados con el Premio Nobel de Medicina en 1978.
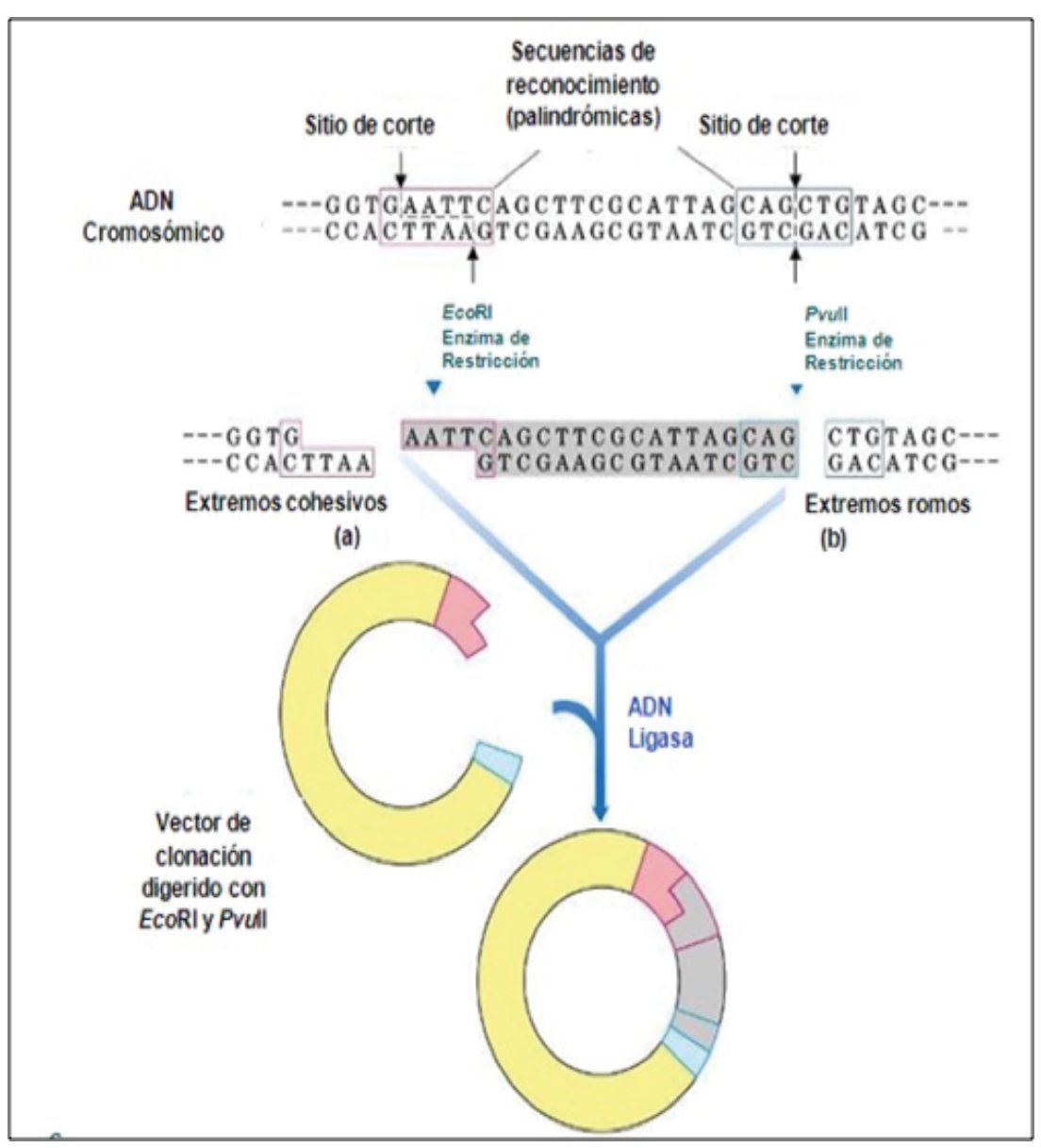
Estas enzimas son conocidas con el término “endonucleasas de restricción”, ya que fueron identificadas por estar implicadas en la restricción del crecimiento de ciertos virus bacterianos (bacteriófagos). La clave para la utilización en el laboratorio de estas “tijeras”, es su estricta especificidad de las secuencias de nucleótidos en el ADN que reconocen y cortan. Una característica de estas secuencias de corte es que son palindrómicas (las dos cadenas de la secuencia de reconocimiento tienen la misma secuencia cuando se leen en sentidos opuestos). Por ejemplo, la bacteria Escherichia coli produce una enzima denominada EcoRI (las enzimas se nombran de acuerdo el género y la especie bacteriana de donde se aislaron), que para realizar el corte reconoce la secuencia 5´-GAATTC-3´. Esta enzima produce cortes escalonados, que dejan cadenas sencillas cortas con extremos “cohesivos” o “pegajosos”. Otras enzimas de restricción como PvuII de la bacteria Proteus vulgaris, realiza los cortes en ambas cadenas del ADN directamente uno frente del otro, generando extremos “romos”.
Si se cortan dos fragmentos de ADN con una de estas “tijeras” de restricción, los cortes realizados en cada fragmento generan extremos que pueden unirse mediante una enzima que se llama ADN ligasa, (que funciona como “pegamento”), realiza la unión de las moléculas de ADN mediante la formación de un enlace fosfodiéster (enlace covalente que une a los nucleótidos).
Vectores o “vehículos” de clonación
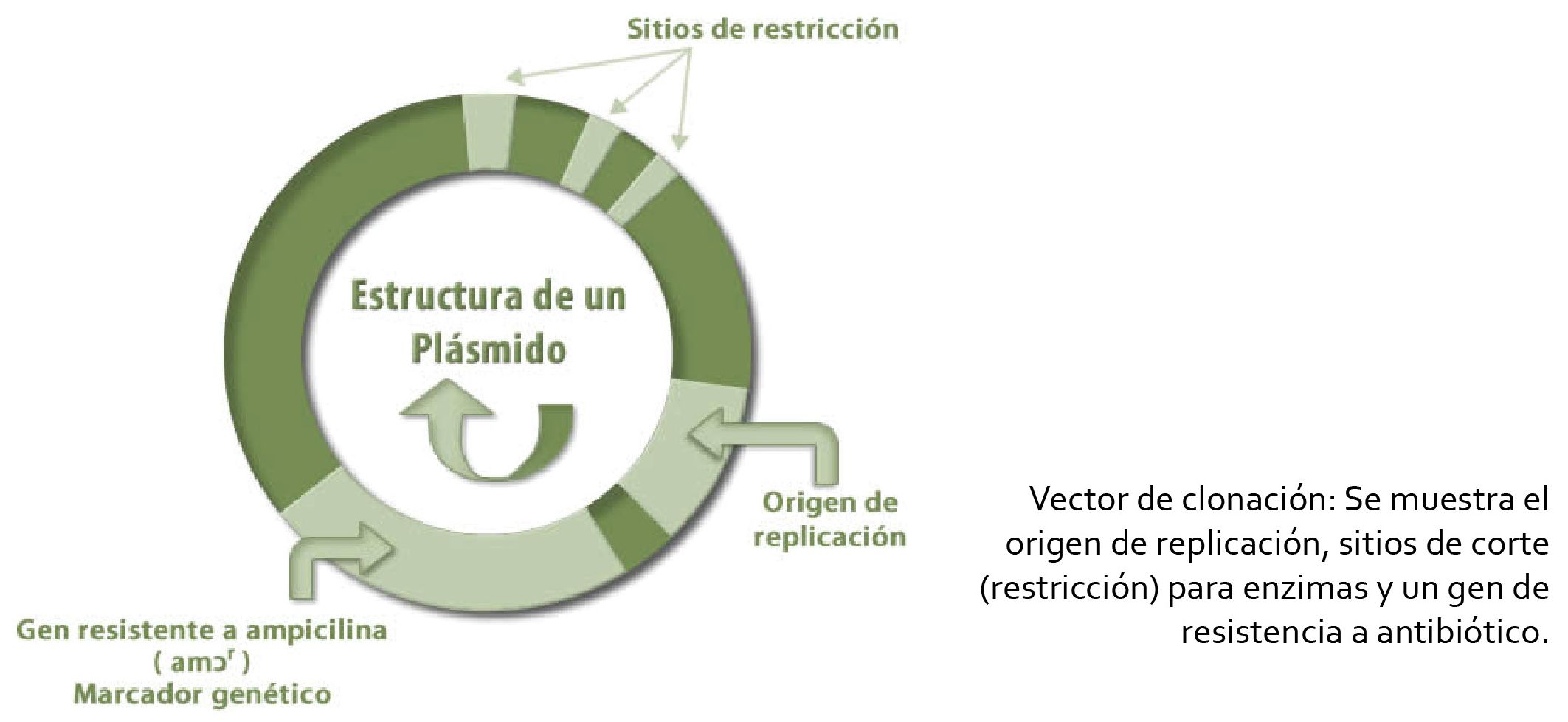
Esta es otra herramienta importante para la clonación de genes, son moléculas de ADN que se replican de manera autónoma y se utilizan para guardar y replicar genes; en ellos es posible insertar ADN exógeno en un sitio de restricción que corta al vector sin afectar su replicación. La mayoría de los vectores proceden de plásmidos, que son moléculas de ADN extracromosómico que se encuentran de manera natural en las bacterias.
Los vectores cumplen con ciertas propiedades que los hacen ideales para almacenar y replicar la información: 1) Poseen un pequeño tamaño que oscila entre 3,000 y 10,000 pb, que permite que su ADN sea manipulado con facilidad; 2) tienen un origen de replicación autónoma, de manera que su replicación en la célula ocurre de manera independiente del control directo del cromosoma; 3) generalmente, tienen alto número de copias (de 500 a 700 copias por célula), lo que genera ADN en gran cantidad; 4) contienen sitios únicos para el corte con varias enzimas de restricción diferentes (entre 10 y 15) que se usan para insertar en esta región (sitio de clonación múltiple) un gen o secuencias de ADN de interés y 5) cuentan con genes que permiten identificar las células que hayan incorporado el vector con la secuencia de ADN exógena, siendo los más utilizados los genes que confieren resistencia a los antibióticos.
En este apartado, es importante una mención especial para el científico mexicano Francisco Bolívar Zapata, cuyo trabajo de investigación pionero (1976), permitió el diseño y construcción de uno de los primeros vectores para la clonación de genes llamado pBR322 (“BR” por Bolívar y Rodríguez, sus creadores).
Introducción del ADN en una célula huésped
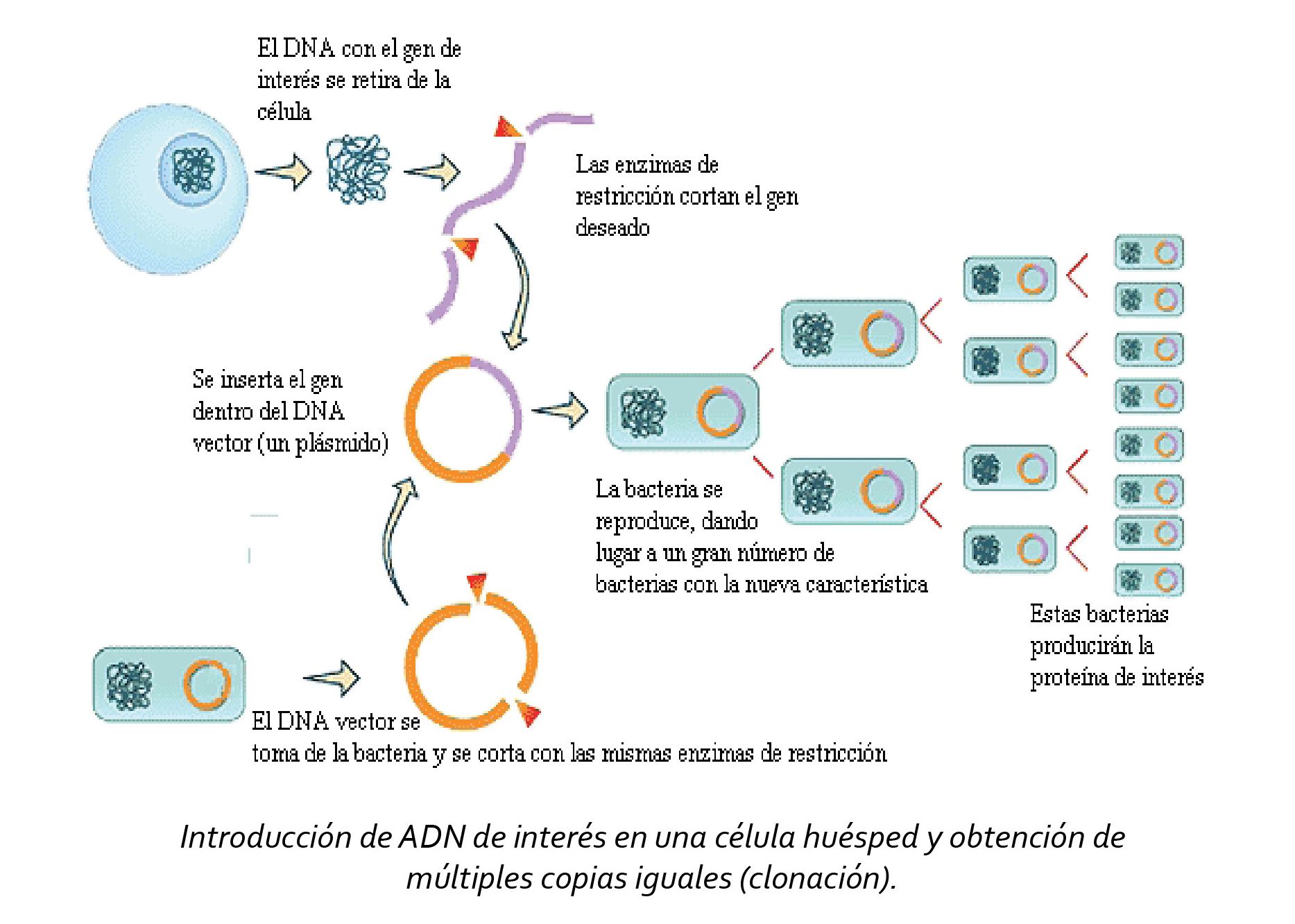
Para la obtención de un gran número de copias de un gen o de una secuencia de interés se requiere una maquinaria celular, por ello hay que introducir el vector en células bacterianas a las que se les llama células huésped. En las bacterias, el vector que tiene origen de replicación autónoma, puede replicarse hasta cientos de veces antes de que la bacteria se divida; al dividirse la bacteria, las nuevas células también contienen el ADN de interés.
El vector es introducido mediante transformación en las células bacterianas; la transformación es un proceso de transferencia de información genética por el cual el ADN es incorporado en una célula receptora y lleva a cabo un cambio genético. Una célula que es capaz de aceptar ADN exógeno y ser transformada se dice que es “competente”.
¿Es posible obtener en el laboratorio células competentes?
Sí, por ejemplo, si las células de E. coli se tratan con soluciones que contienen altas concentraciones de iones calcio (CaCl2) y se hacen cambios rápidos de temperatura (de 42° a 4°C) de incubación por uno o dos minutos, con ello se modifica la permeabilidad de la membrana, lo que causa que el ADN penetre en la célula. Otro mecanismo para introducir ADN a células bacterianas es mediante electroporación, en el que las células se exponen a breves pulsos eléctricos, del orden de los milisegundos (1 ms=0.001s) de alto voltaje, lo que genera poros en la membrana permitiendo la entrada del ADN a la célula.
¿Cómo identificar el gen de interés?
Después de transformar a las células huésped con el vector que tiene el gen o secuencia de interés, es necesario identificar las células que lo contienen. Esto es debido a que la frecuencia de transformación no es del 100% de las células, ya que sólo aproximadamente 1 de cada 10,000 células incorpora con éxito el ADN exógeno. Sin embargo, como el tamaño de la población de células receptoras es muy grande (100,000 a 100,000,000 células), aún a estas bajas frecuencias, pueden detectarse fácilmente en el cultivo.
Para ello, las células huésped se crecen en una placa de cultivo donde formarán colonias (acúmulos de células bacterianas cuando se les cultiva en el laboratorio). Hay que considerar que las células de cada una de las colonias provienen de una sola célula inicial, por lo que todas las células de la colonia y los plásmidos que contienen son genéticamente idénticas o clones.
La colonia de interés se puede identificar a través del marcador de selección del vector, que como se había señalado con anterioridad, uno de los más utilizados es la resistencia a un antibiótico. Con esto en mente, en el medio de cultivo se agrega el antibiótico de selección y sólo crecerán aquellas colonias que sean resistentes a él y, por tanto, que porten el ADN del vector con el gen que genera la resistencia a dicho antibiótico y también el gen o secuencia de interés clonado.
En este momento se considera que la información genética se ha clonado en una célula bacteriana, la cual puede almacenarse y mantener la información por mucho tiempo.
Lo anterior es el resultado de estas dos herramientas principales: las tijeras y los vectores moleculares, utilizados en los laboratorios de biología molecular de investigación o de docencia.
Saber más:
Clonación: vectores. 2012. Portal Académico-CCH.
http://portalacademico.cch.unam.mx/alumno/biologia1/unidad3/ingenieriagenetica/ADNrecombinante/clonacionVectores
Brock, 2009. Biología de los microorganismos. 12 Ed. 344-351 pp.
La M. en C. Martha Patricia Chávez Moctezuma es Técnico Académico Titular del Laboratorio de Microbiología del Instituto de Investigaciones Químico-Biológicas de la Universidad Michoacana de San Nicolás de Hidalgo.
